- Recrutements
- Contacts
- Annuaires
- Choix du langage :
Dernières publications
948.
- titre
- Do suspended particles matter for wastewater-based epidemiology?
- auteur
- Gauthier Bernier-Turpin, Régis Moilleron, Chloé Cenik, Fabrice Alliot, Sabrina Guérin-Rechdaoui, Thomas Thiebault
- article
- , In press, 280, pp.123543. ⟨10.1016/j.watres.2025.123543⟩
- titre
- Stock and vertical distribution of microplastics and tire and road wear particles into the soils of a high-traffic roadside biofiltration swale
- auteur
- Max Beaurepaire, Tiago de Oliveira, Johnny Gasperi, Romain Tramoy, Mohamed Saad, Bruno Tassin, Rachid Dris
- article
- , 2025, 373, pp.126092. ⟨10.1016/j.envpol.2025.126092⟩
- titre
- Assessing water quality restoration measures in Lake Pampulha (Brazil) through remote sensing imagery
- auteur
- Alexandre Assunção, Talita Silva, Lino de Carvalho, Brigitte Vinçon-Leite
- article
- , 2025, ⟨10.1007/s11356-025-35914-6⟩
- titre
- How to monitor and forecast microbiological quality in bathing sites in urban water bodies? The La Villette study site (Paris)
- auteur
- Arthur Guillot - Le Goff, Natalia Angelotti de Ponte Rodrigues, Rémi Carmigniani, Brigitte Vinçon-Leite
- article
- , 2025, TSM 12/2024, pp.219-228. ⟨10.36904/tsm/202412219⟩
- titre
- Modelling evapotranspiration in urban green stormwater infrastructures: Importance of sensitivity analysis and calibration strategies with a hydrological model
- auteur
- Ahmeda Assann Ouédraogo, Emmanuel Berthier, Jérémie Sage, Marie-Christine Gromaire
- article
- , 2025, 185, pp.106319. ⟨10.1016/j.envsoft.2025.106319⟩
Offre de thèse 2023 : Relier la pression chimique à ses effets sur la biodiversité dans les milieux aquatiques en couplant l’analyse non ciblée avec l’ADN environnemental à l’aide d’outils numériques
publié le
1. Contexte
La qualité des milieux aquatiques et de la ressource en eau peut être appréhendée du point de vue de sa contamination chimique, en particulier par des contaminants émergents, ou de la biodiversité qu’elle accueille. La présence de ces contaminants est néfaste pour la santé des écosystèmes avec des effets sur les organismes aquatiques et une perte de biodiversité.
Afin de caractériser cette contamination chimique dans les milieux aquatiques, des méthodes d’analyse non ciblée par spectrométrie de masse haute résolution (HRMS) ont été développées. Au Leesu, deux thèses ont été menées sur cette thématique et ont permis d’une part de développer la méthode analytique (Huynh et al. 2021) et les outils informatiques de traitement des données (Sade et al. 2022), et d’autre part d’appliquer ces méthodes à différentes eaux urbaines afin d’évaluer la variabilité spatio-temporelle de la contamination.
En parallèle, l’étude de l’ADN environnemental (ADNe), dans lequel les signaux des macro- et micro-organismes de l’environnement sont récupérés à partir d’échantillons d’eau est de plus en plus utilisée pour la surveillance de la biodiversité des milieux. Ces méthodes permettent de détecter les espèces aussi bien, voire mieux, que les méthodes traditionnelles d’étude de biodiversité, en particulier dans l’environnement aquatique qui peut intégrer des informations sur la biodiversité de tout le bassin versant.
Les progrès techniques qui permettent aujourd’hui d’enregistrer des empreintes chimiques par des approches non ciblées, ou de mesurer l’ADN environnemental pour étudier la biodiversité, fournissent d’énormes quantités de données. Le défi consiste à mettre en place les outils d’analyse biologique et chimique multivariée pour faciliter l’utilisation de ces données. L’utilisation d’outils numériques comme le machine learning se développe pour traiter et interpréter les spectres HRMS de contaminants dans les eaux, de même que dans le domaine de l’ADN environnemental pour extraire des indicateurs de biodiversité ou expliquer les variabilités spatio-temporelles.
Pour l’instant ces deux approches n’ont pas été couplées. Or les mélanges complexes de produits chimiques doivent être pris en compte en même temps que leurs effets complexes et leurs impacts sur les écosystèmes. Le couplage entre l’empreinte chimique des contaminants et l’ADNe qui mesure la biodiversité est un enjeu majeur pour la surveillance de la qualité des milieux récepteurs. Des essais de couplage entre ADNe et hydrodynamique ont été tentés mais pour l’instant les études couplant spectres HRMS et ADNe sont rares.
L’objectif général de ce projet de thèse est donc d’évaluer la possibilité d’interpréter conjointement les empreintes chimiques de contaminants dans les eaux avec les informations sur la biodiversité données par l’ADNe en développant et appliquant des méthodes numériques de type machine learning.
2. Cadre et partenaires du projet
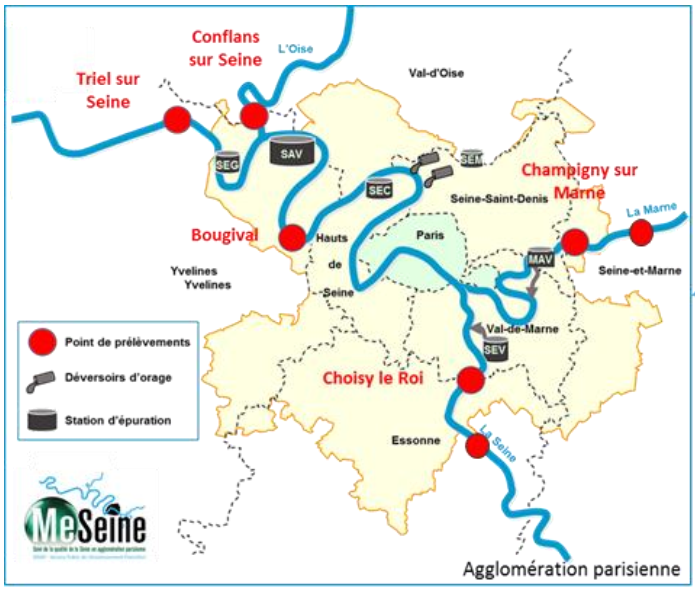
Cette thèse s’insère dans les actions de recherche du Leesu en collaboration avec le programme MeSeine Innovation porté par le Service Public de l’Assainissement Parisien (SIAAP) dont un des objectifs est d’améliorer le suivi des masses d’eau en couplant différentes approches hydrologiques, chimiques et biologiques. Des campagnes de prélèvements sur 4 à 7 sites répartis entre la Seine, la Marne et l’Oise sont réalisées régulièrement (figure 1).
Les analyses de contaminants émergents s’appuient sur les instruments analytiques de la plateforme Prammics (OSU Efluve), en particulier les instruments de chromatographie en phase liquide (Waters Vion – UPLC-IMS-QTOF et Shimadzu HPLC Fraction collector). L’UPLC-IMS-QTOF est un instrument de HRMS équipé d’une séparation par mobilité ionique (IMS). L’ADNe sera mesuré par un prestataire.
3. Objectifs
Les objectifs de la thèse sont les suivants :
- Identifier pour chaque type de données (HRMS et ADNe) des traceurs ou indicateurs d’intérêt à partir de la littérature scientifique et automatiser leur traitement.
- Développer des méthodes de traitement des données pour coupler les données HRMS et d’ADNe en utilisant des outils statistiques avancés ou numériques de type machine learning.
- Proposer une stratégie d’échantillonnage pertinente pour acquérir les deux types de données (HRMS et ADNe) sur le réseau MeSeine et effectuer un suivi régulier des différents points d’échantillonnage.
- Suivre et interpréter le devenir spatio-temporel de molécules/marqueurs d’intérêt identifiés précédemment (suivi à différentes fréquences : saisonnières, hebdomadaires, journalières et le long des différents points de prélèvement en Seine)
4. Laboratoire d’accueil
La thèse se déroulera au Laboratoire Eau Environnement et Systèmes Urbains (LEESU - UMR MA102 (Université Paris-Est Créteil et École des Ponts ParisTech), principalement sur le site de la Maison des Sciences de l’Environnement à Créteil (61 avenue du Général de Gaulle, Créteil). Une partie de la thèse pourra être menée au sein des locaux du SIAAP à Colombes.
4. Profil recherché
Formation de niveau M2 ou ingénieur en bioinformatique/biostatistiques, chimie analytique ou chimie de l’environnement :
- Compétences en statistiques (régressions, classification automatique discriminante, arbres de décision…) et analyse de données, machine learning
- Compétences en programmation, utilisation de langages de programmation pour le traitement de données (data science) (R, python…)
- Compétences en chimie analytique (spectrométrie de masse, métabolomique) et/ou biologie moléculaire
- Des connaissances en sciences de l’environnement (polluants, qualité des eaux et notions de biodiversité) sont bienvenues
- Aisance dans la rédaction et bon niveau d’anglais
5. Contacts et modalités de candidature
Encadrement
- Adèle Bressy, chargée de recherche HDR de l’École des Ponts, chimie de l’environnement, hydrologie (adele.bressy[at]enpc.fr)
- Julien Le Roux, maître de conférences UPEC, chimie analytique, outils statistiques et numériques, chimie de l’environnement (julien.le-roux[at]u-pec.fr)
- My Dung Jusselme, maîtresse de conférence UPEC, biologie moléculaire, microbiologie environnementale
- Sabrina Guérin, responsable innovation au SIAAP
Candidature
Envoyer CV, lettre de motivation et contact de référent.e.s scientifiques à adele.bressy[at]enpc.fr et julien.le-roux[at]u-pec.fr.
Offre de thèse (fichier PDF)









 Productions scientifiques
Productions scientifiques Moyens techniques et équipements
Moyens techniques et équipements Expertise et disciplines
Expertise et disciplines


