- Recrutements
- Contacts
- Annuaires
- Choix du langage :
Dernières publications
935.
- titre
- Assessing water quality restoration measures in Lake Pampulha (Brazil) through remote sensing imagery
- auteur
- Alexandre Assunção, Talita Silva, Lino de Carvalho, Brigitte Vinçon-Leite
- article
- Environmental Science and Pollution Research, 2025, ⟨10.1007/s11356-025-35914-6⟩
- titre
- Do suspended particles matter for wastewater-based epidemiology?
- auteur
- Gauthier Bernier-Turpin, Régis Moilleron, Chloé Cenik, Fabrice Alliot, Sabrina Guérin-Rechdaoui, Thomas Thiebault
- article
- Water Research, In press, 280, pp.123543. ⟨10.1016/j.watres.2025.123543⟩
- titre
- Plastic debris dataset on the Seine riverbanks: up to 38 000 pre-production plastic pellets reported per square meter
- auteur
- Romain Tramoy, Laurent Colasse, Johnny Gasperi, Bruno Tassin
- article
- Data in Brief, 2025, pp.111735. ⟨10.1016/j.dib.2025.111735⟩
- titre
- La persistance des champs d’épandage d’eaux usées de l’agglomération parisienne au cours du second XXe siècle
- auteur
- Etienne Dufour
- article
- Métropolitiques, 2025, ⟨10.56698/metropolitiques.2174⟩
- titre
- Stock and vertical distribution of microplastics and tire and road wear particles into the soils of a high-traffic roadside biofiltration swale
- auteur
- Max Beaurepaire, Tiago de Oliveira, Johnny Gasperi, Romain Tramoy, Mohamed Saad, Bruno Tassin, Rachid Dris
- article
- Environmental Pollution, 2025, 373, pp.126092. ⟨10.1016/j.envpol.2025.126092⟩
Contaminants microbiologiques
publié le , mis à jour le
Contaminants microbiologiques
Le laboratoire de microbiologie dispose des appareils nécessaires à la réalisation d’analyses en microbiologie classique et en biologie moléculaire.
- Matériel de terrain
Afin de recueillir de l’eau dans différents compartiments du milieu naturel, nous avons fabriqué un système permettant d’échantillonner le neuston (couche superficiel d’une surface liquide) grâce à une grille fine. Nous utilisons également une bouteille Niskin pouvant prélever de l’eau à différentes profondeurs.
Des batteries associées à un convertisseur fournissent l’électricité nécessaire au fonctionnement de pompes pour la filtration sur le terrain.

- Microbiologie classique
- Ensemencement
Les hottes à flux laminaire (BioII, ADS Laminaire) nous permettent de travailler dans des conditions stériles afin de ne pas contaminer nos échantillons et de protéger le manipulateur. Les analyses menées consistent à l’ensemencement de microplaques contenant un milieu spécifique permettant de dénombrer deux indicateurs de contaminations fécales, Escherichia coli et les entérocoques intestinaux (normes). Ces indicateurs bactériens nous permettent d’apprécier la qualité microbiologique des eaux analysées au laboratoire, qu’elles proviennent du milieu naturel ou de station d’épuration. La lecture des microplaques se fait sous lumière ultraviolette qui révèle par fluorescence les puits positifs.
Nous pouvons également réaliser des ensemencements sur milieu gélosé ou liquide afin de cultiver des souches d’intérêt.

Le laboratoire possède également deux lecteurs de microplaque dans la lumière visible (Anthos Reader, Labgen) et en fluorescence (Fluoroscan, Thermo).
-
- Microscopie en fluorescence (BH2, Olympus) et microscopie inversée (Primovert, Zeiss et camera AxioCam ERc 5S)
Les échantillons fixés au paraformaldéhyde et colorés au DAPI sont filtrés sur membrane de porosité 0.22µm afin de dénombrer les cellules totales (bactéries et Archées) par microscopie en fluorescence.
Les microalgues sont dénombrées par microscopie inversée.
- Préparation des échantillons
Des systèmes de filtration ont été montés afin de concentrer sur des filtres cylindriques de porosité 0.22µm (Millipore, Sterivex GP) les bactéries présentes dans les eaux.
Une centrifugeuse gros volumes (Avanti JE, Beckman) permet de concentrer les échantillons et donc de récupérer le culot cellulaire des matrices liquides.
- Biologie moléculaire
- Extraction d’ADN
Grâce à des kits spécifiques (Spin Kit for Soil, PMBiomedical) et à la mise au point de protocoles adaptés à nos matrices, nous pouvons extraire l’ADN des cellules bactériennes présentes dans les échantillons analysés après filtration. Les quantités extraites sont estimées par spectrophotométries (UviLine 9400, Secoman).
Après précipitation à l’éthanol, les culots sont séchés dans un évaporateur rotatif (MiVac, Bioblock).
-
- PCR (Polymerisation Chain Reaction)
Le laboratoire est pourvu d’un poste dédié à la PCR (pré-PCR et post-PCR) équipé de deux thermocycleurs.
Le premier thermocycleur (T1, Biometra) sert
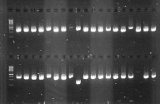
à l’amplification de fragments spécifiques d’ADN comme par exemple le gènes de l’ARN ribosomal 16S qui peut ensuite être séquencés afin d’identifier les individus d’une population (clonage séquençage ou séquençage haut débit). Nous utilisons un système d’acquisition d’image (MS geldoc, dutscher) pour vérifier la bonne amplification des fragments d’intérêt.
Le second est un thermocycleur en temps réel (iQ Biorad) qui sert à estimer la quantité d’individu dans un échantillon par amplification d’une séquence spécifique marquée par un fluorophore. Le laboratoire dispose d’une méthode de quantification des mycobactéries non tuberculeuses (genre Mycobacterium) par amplification du gène atpE (Radomski et al., 2013). Cette technique de PCR en temps réel permettra également la détection de pathogènes d’intérêt tels que les salmonelles et Campilobacter.









 Productions scientifiques
Productions scientifiques Moyens techniques et équipements
Moyens techniques et équipements Expertise et disciplines
Expertise et disciplines


